虫媒黄病毒成员众多,包括寨卡病毒(Zika virus,ZIKV)、登革病毒、西尼罗病毒、黄热病毒等在全球范围内广泛分布,频繁暴发,严重威胁人类健康。虫媒黄病毒传播方式相近,基因组结构类似,然而其导致的临床表现却存在很大差异,尤其是寨卡病毒可高效穿越血胎屏障,导致新生儿小头畸形等严重出生缺陷疾病,影响人类生殖安全。2016年南美的寨卡大流行被世界卫生组织宣布为"全球关注的突发公共卫生事件"。
与其他虫媒黄病毒相比,寨卡病毒对神经前体细胞(neural progenitor cells,NPCs)具有很强的嗜性,但随着神经前体细胞的进一步分化,其感染性显著降低[1]。科学界对寨卡病毒这种显著区别于其他虫媒黄病毒的特异组织嗜性特性一直缺乏合理解释。传统的观点认为,病毒对特定组织细胞的嗜性主要与病毒编码蛋白有关。如早期研究发现,寨卡病毒prM蛋白单个氨基酸突变S139N可显著增强其对神经前体细胞的感染能力[2],E21K突变可显著增强病毒对小鼠神经毒力和神经侵袭力[3]。而病毒基因组RNA主要参与调控复制和翻译,与病毒的组织细胞嗜性无直接关联。
2023年2月28日,中国科学院生物物理研究所方显杨课题组与军事医学研究院秦成峰课题组在《Nature Communications》上合作发表了题为"Zika virus RNA structure controls its unique neurotropism by bipartite binding to Musashi-1"的研究论文。研究发现寨卡病毒基因组中存在特殊的RNA高级结构,可通过与宿主蛋白MSI1结合进而调控病毒的神经嗜性,从RNA层面揭示了寨卡病毒独特的神经嗜性调控机制,也为RNA的结构和功能研究提供了全新的方向。
Musashi-1(MSI1)蛋白是一种重要的神经发育调节蛋白,在神经前体细胞中高丰度表达。早期研究发现,MSI1可与寨卡病毒基因组的3'UTR结合并促进病毒复制[4],但具体作用机制并不清楚。作为典型的RNA结合蛋白,MSI1可结合多种宿主mRNA,其特异识别基序为A/GU(1-3)AG。研究人员首先通过序列分析发现,寨卡病毒3'UTR上存在3个符合A/GU(1-3)AG特征的位点,分别分布于xrRNA2,DB12和3'SL区(图1a)。进一步生化分析和反向遗传学实验证实,3个区域中只有xrRNA2中的AUAG基序呈现出很强的MSI1结合能力(图1b),而且MSI-1的两个RRM同时为高亲和力结合所必需(图1c)。
xrRNA是一类源自虫媒黄病毒基因组3'UTR的RNA,其形成的独特的环状结构,可抵抗宿主5'→3'核糖核酸外切酶Xrn1降解的功能[5]。寨卡病毒xrRNA2是寨卡病毒第2个xrRNA,其结构尚未被解析。在试图解析xrRNA2与MSI1复合物晶体结构的过程中,研究人员意外发现xrRNA2中的P2四核苷酸环(tetraloop)对于MSI1的结合同样至关重要。包含AGAA序列的P2属于经典的AGNN形式的tetraloop,可与MSI1高效结合;而突变为GAAA、GAGA和GCAA等GNRA类型的tetraloop突变则显著降低了MSI1的结合能力。进一步的突变分析和反向遗传学实验证实,xrRNA2中P2只有维持AGNN类型才能在神经前体细胞中促进寨卡病毒的增殖。也就是说,xrRNA2中AUAG基序和AGNN类型的tetraloop结构共同负责与MSI1结合,进而决定了寨卡病毒在神经前体细胞中的高效复制(图1d)。
更为重要的是,研究者比对了所有虫媒黄病毒基因组的3'UTR,发现只有寨卡病毒同时具备AUAG基序和AGNN类型的tetraloop结构,其他一些虫媒黄病毒成员的3'UTR存在AUAG基序,但缺乏AGNN类型的tetraloop结构,功能试验也证实这些病毒的RNA均不能与MSI1有效结合;敲低MSI1蛋白只对寨卡病毒复制有抑制作用,而对其它病毒没有影响。进一步通过反向遗传学技术在登革病毒3'UTR中引入AUAG基序和AGNN类型的tetraloop结构,结果发现,拯救获得的重组登革病毒病毒不仅获得了与MSI1高效结合的能力,对神经前体细胞的感染能力也显著增加。因此,寨卡病毒xRNA2中AUAG基序和AGNN类型的tetraloop结构及其形成的特定结构,共同决定了寨卡病毒独特的神经嗜性,使之区别于其他虫媒黄病毒。
为解析xrRNA2特异性结合MSI1的相互作用模式,研究者通过整合小角X射线散射(SAXS)、氢氘交换质谱(HDX-MS)、定点突变、结构预测与计算模拟等多种结构生物学研究方法,得到了xrRNA2与MSI1相互作用的结构模型。结果表明,MSI1蛋白的两个RNA识别亚基(RRM1、RRM2)通过错位平行的方式分别与xrRNA2的两个位点(pMBS1、P2 tetraloop)结合(图1e)。xrRNA2所具备的三级结构特征,包括整体的折叠、局部的结构以及一级序列,是其可以特异性的与MSI1相互作用的原因, 相比之下,宿主mRNA往往通过A/GU(1-3)AG识别基序与MSI1相互作用。这些模型特征可以帮助我们更好的理解寨卡病毒依靠其独特的RNA结构识别MSI1并利用其帮助病毒复制的机理。
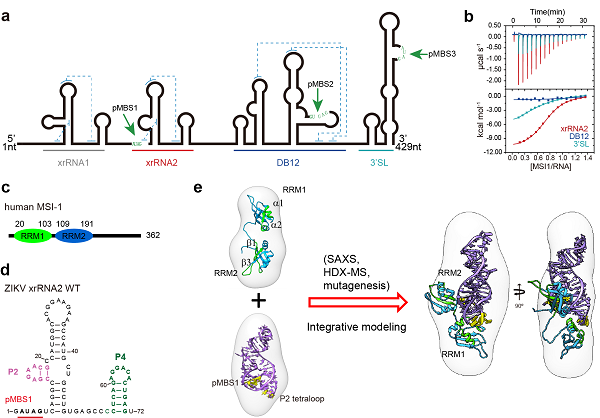
图1. 寨卡病毒xrRNA2特异性识别神经发育蛋白MSI-1以调控其神经嗜性的分子机制。a) 寨卡病毒3'UTR存在3个潜在的MSI-1结合位点(MBS), b) ITC实验确认xrRNA2为MSI-1的首要结合元件,c) MSI-1的结构示意图,其包含两个RRM,d) xrRNA2的二级结构图及MSI-1的两个结合位点,e) 基于多方法联用的xrRNA2与MSI-1的RRM12的整合结构模型。
综上,该项研究不仅鉴定出决定寨卡病毒神经嗜性的关键RNA高级结构元件,回答了领域内长期悬而未解的难题;也为下一代疫苗和抗病毒药物设计提供了重要靶标,为病毒RNA的结构与功能研究提供了新的方向。
军事医学研究院博士后陈祥、清华大学生命学院已毕业博士生王岩和2017级博士生徐钟河为本文的共同第一作者。军事医学研究院秦成峰研究员和中国科学院生物物理研究所方显杨研究员为本文的共同通讯作者。研究工作得到了国家重点研发计划、国家杰出青年基金等项目的支持。
文章链接:https://doi.org/10.1038/s41467-023-36838-w
参考文献
1. Li C, Xu D, Ye Q, Hong S, Jiang Y, Liu X, Zhang N, Shi L, Qin CF, Xu Z: Zika Virus Disrupts Neural Progenitor Development and Leads to Microcephaly in Mice. Cell Stem Cell 2016, 19:672.
2. Yuan L, Huang XY, Liu ZY, Zhang F, Zhu XL, Yu JY, Ji X, Xu YP, Li G, Li C, et al: A single mutation in the prM protein of Zika virus contributes to fetal microcephaly. Science 2017, 358:933-936.
3. He MJ, Wang HJ, Yan XL, Lou YN, Song GY, Li RT, Zhu Z, Zhang RR, Qin CF, Li XF: Key Residue in the Precursor Region of M Protein Contributes to the Neurovirulence and Neuroinvasiveness of the African Lineage of Zika Virus. J Virol 2023:e0180122.
4. Chavali PL, Stojic L, Meredith LW, Joseph N, Nahorski MS, Sanford TJ, Sweeney TR, Krishna BA, Hosmillo M, Firth AE, et al: Neurodevelopmental protein Musashi-1 interacts with the Zika genome and promotes viral replication. Science 2017, 357:83-88.
5. Akiyama BM, Laurence HM, Massey AR, Costantino DA, Xie X, Yang Y, Shi PY, Nix JC, Beckham JD, Kieft JS: Zika virus produces noncoding RNAs using a multi-pseudoknot structure that confounds a cellular exonuclease. Science 2016, 354:1148-1152.
(供稿:方显杨研究组)
 附件下载:
附件下载: